Vea También
 |
| Los investigadores planean ahora dotarlo de un acoplamiento e instalarlo como accionamiento en nanomáquinas complejas. Crédito: Unsplash/CC0 Public Domain |
La ingeniería molecular pretende desarrollar unidades funcionales modulares que puedan utilizarse en el diseño ascendente de nanoensamblajes capaces de llevar a cabo tareas exigentes. Estos sistemas necesitan nanomotores que consuman combustible y puedan empujar activamente a los seguidores pasivos aguas abajo. Salvo raras excepciones, el movimiento browniano impulsa la mayoría de los motores moleculares artificiales, pero las fuerzas que se crean suelen ser no dirigidas e insuficientes para transmitirse eficazmente a los componentes pasivos de segundo nivel. En consecuencia, aún no se han desarrollado sistemas nanoescalares eficaces de conductor-seguidor accionados por combustible químico.
Un nuevo estudio de un equipo internacional de científicos presentó una nanomáquina de ADN, un novedoso tipo de nanomotor hecho de ADN. Puede realizar movimientos pulsantes y está propulsada por un mecanismo inteligente. La idea de los investigadores es instalarla como propulsor en intrincadas nanomáquinas tras dotarla de una conexión.
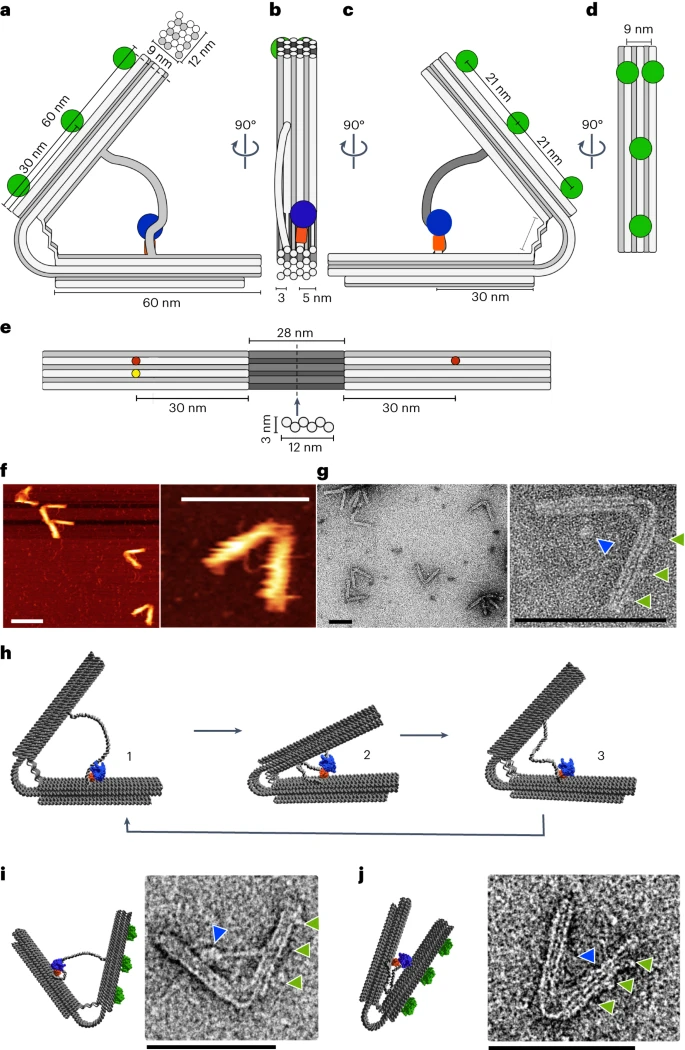
Los científicos utilizaron las herramientas de modelado informático del grupo para comprender mejor el diseño y funcionamiento de este nanomotor de ballesta. Casi 14.000 nucleótidos, los bloques estructurales fundamentales del ADN, componen la estructura.
Petr Šulc, profesor adjunto de la Facultad de Ciencias Moleculares y del Centro de Diseño Molecular y Biomimética de la Universidad Estatal de Arizona, declaró: "Ser capaces de simular el movimiento en una nanoestructura tan grande sería imposible sin oxDNA, el modelo informático que nuestro grupo utiliza para el diseño y la concepción de nanoestructuras de ADN."
"Es la primera vez que se consigue diseñar con éxito un motor nanotecnológico de ADN accionado químicamente. Nos entusiasma que nuestros métodos de investigación hayan podido ayudar a estudiarlo, y estamos deseando construir nanodispositivos aún más complejos en el futuro."
Este innovador motor funciona de forma similar a un ejercitador de manos que fortalece el agarre cuando se utiliza con frecuencia. El motor, sin embargo, es alrededor de un millón de veces más pequeño. En forma de V, dos empuñaduras están unidas por un muelle.
El profesor Michael Famulok (director del proyecto), de la Universidad de Bonn (Alemania), explica: "En un entrenador de fuerza de agarre, se aprietan las empuñaduras contra la resistencia del muelle. Cuando sueltas el agarre, el muelle empuja las empuñaduras a su posición original. Nuestro motor utiliza un principio muy similar. Pero las empuñaduras no se aprietan, sino que se tiran".
Según los expertos, sin un mecanismo específico no habría plantas ni criaturas en la Tierra. En cada célula hay una pequeña biblioteca. Contiene las instrucciones para cada tipo de proteína que una célula necesita para funcionar. La célula solicita una copia del plano adecuado para fabricar un tipo específico de proteína. Las ARN polimerasas son las enzimas encargadas de crear esta transcripción.
Las largas cadenas de ADN constituyen el modelo inicial. A estas hebras les siguen las ARN polimerasas, que copian letra a letra la información allí almacenada.
Famulok explica: "Tomamos una ARN polimerasa y la unimos a una de las asas de nuestra nanomáquina. Cerca, también colamos una cadena de ADN entre las dos asas. La polimerasa se agarra a esta cadena para copiarla. Tira de la cadena y la sección no transcrita se hace cada vez más pequeña. Esto tira del segundo asa poco a poco hacia el primero, comprimiendo el muelle simultáneamente".
Justo antes de terminar, la cadena de ADN entre las asas lleva un conjunto específico de letras. Esta llamada secuencia de terminación ordena a la polimerasa que libere el ADN de su agarre. Una vez más relajado, el muelle puede separar las asas. Como resultado, la copiadora molecular puede iniciar un nuevo proceso de transcripción porque la secuencia de inicio de la cadena está ahora cerca de la polimerasa. El proceso continúa entonces.
Mathias Centola, que forma parte del grupo de investigación dirigido por Famulok y realizó gran parte de los experimentos, afirmó: "De este modo, nuestro nanomotor realiza una acción pulsante".
Como todos los motores, éste necesita energía para funcionar. Se la da la "sopa de letras" con la que la polimerasa fabrica los transcritos. Cada una de estas letras, o nucleótidos en la jerga científica, tiene una pequeña cola de tres grupos fosfato, o un trifosfato. La polimerasa debe eliminar dos grupos fosfato para añadir una nueva letra a una frase ya existente. Como resultado, se libera energía que puede utilizarse para conectar las letras.
Según Famulok, "nuestro motor utiliza así trifosfatos de nucleótidos como combustible. Sólo puede seguir funcionando cuando se dispone de un número suficiente de ellos".
A continuación, los científicos demostraron que el motor puede combinarse con otras estructuras. Debería ser capaz de hacer cosas como serpentear por una superficie, de forma parecida a un gusano que se empuja a sí mismo por una rama de forma única.
Según Famulok, "también estamos planeando fabricar un tipo de embrague que nos permita utilizar la potencia del motor sólo en determinados momentos y, de lo contrario, dejarlo al ralentí". A largo plazo, el motor podría convertirse en el corazón de una nanomáquina compleja. Sin embargo, queda mucho trabajo por hacer antes de llegar a esta fase".
En concreto, el grupo desarrolla nuevos modelos multiescala para crear y modelizar nanoestructuras de ADN y ARN y dispositivos para examinar las interacciones entre biomoléculas.
Šulc afirmó: "Al igual que las máquinas complejas de nuestro uso cotidiano -aviones, coches y chips en electrónica- requieren sofisticadas herramientas de diseño asistido por ordenador para asegurarse de que cumplen la función deseada, existe una necesidad acuciante de acceder a tales métodos en las ciencias moleculares."
Fuentes, créditos y referencias: