Vea También

El genoma es la secuencia completa de ADN de un organismo, que suele ser casi idéntica entre individuos de una misma especie. Desde una perspectiva evolutiva, puede considerarse un sello de identidad, pues contiene la información genética única de cada organismo. A su vez, las mutaciones que alteran esta secuencia pueden originar cambios biológicos que, en algunos casos, dan lugar a la aparición de nuevas especies.
En las últimas décadas, la secuenciación de genomas de miles de especies nos ha permitido comprender mejor cómo la evolución ha moldeado la extraordinaria diversidad de la vida en la Tierra. Uno de los hallazgos más reveladores es que la pérdida de genes puede desempeñar un papel tan importante como su adquisición en la evolución de las especies.
Menos es más: la pérdida génica en la evolución
Aunque intuitivamente asociamos la pérdida de genes con una reducción de funciones, la hipótesis de “menos es más” propone que en algunos casos las pérdidas pueden conferir ventajas evolutivas.
Existen múltiples ejemplos: en humanos, la pérdida de ciertos receptores celulares otorga resistencia a enfermedades como la malaria o el VIH. En colibríes, la pérdida de genes del metabolismo energético y de la musculatura facilitó la evolución de su característico vuelo estacionario.
Sin embargo, la pérdida de un gen no solo implica la desaparición de su función, sino que también afecta la evolución de otros elementos del genoma con los que antes interactuaba. Aunque aún no se comprenden del todo los mecanismos y consecuencias de este proceso, su estudio es esencial para entender cómo la pérdida génica ha moldeado la biodiversidad.
La secuenciación masiva de genomas ha revelado que la pérdida génica ha ocurrido con distinta intensidad en cada rama del árbol de la vida, siendo especialmente frecuente en ciertos grupos de seres vivos. En algunos organismos, la eliminación de genes parece haber sido un motor evolutivo clave, convirtiéndolos en modelos ideales para investigar este fenómeno.
Oikopleura dioica: un perdedor exitoso
Oikopleura dioica, un diminuto organismo planctónico presente en mares de todo el mundo, se ha convertido en un modelo clave en nuestro laboratorio en la Sección de Genética y el Instituto de Investigación de la Biodiversidad (IRBio) de la Universidad de Barcelona para estudiar la pérdida génica. Pertenece a las apendicularias, un subgrupo de tunicados, los parientes más cercanos de los vertebrados.
A lo largo de su evolución, el genoma de O. dioica se ha reducido drásticamente hasta quedar en menos de 70 megabases, unas 50 veces más pequeño que el humano. Esta reducción ha implicado una pérdida masiva de genes, muchos de ellos vitales en otros animales, lo que convierte a O. dioica en un verdadero enigma evolutivo.
Rompecabezas genético
Entre sus pérdidas más notables, destaca la de la vía de señalización por ácido retinoico (RA), un derivado de la vitamina A crucial para el desarrollo embrionario. Esto plantea preguntas clave: ¿cómo logra O. dioica desarrollarse sin RA? ¿qué impacto ha tenido esta pérdida en la evolución de otros mecanismos reguladores del desarrollo?
Para responder a esta cuestión, hemos analizado otra vía: la de los Factores de Crecimiento de Fibroblastos (FGF). Hace unos 600 millones de años, en el ancestro común de tunicados y vertebrados, las vías de RA y FGF se equilibraban mutuamente para definir los ejes corporales de los embriones. Ese equilibrio se ha mantenido en todos sus descendientes… excepto en las apendicularias. La desaparición de la vía de RA en O. dioica convierte así a la vía de FGF en un modelo ideal para estudiar los efectos evolutivos de la pérdida génica.
Nuestra investigación ha revelado que, aunque O. dioica conserva la vía de FGF, esta también ha sufrido pérdidas masivas. De las ocho subfamilias originales heredadas del ancestro común de los cordados, solo sobreviven dos: Fgf9/16/20 y Fgf11/12/13/14. La primera agrupa factores de señalización extracelulares cruciales para el desarrollo embrionario, mientras que la segunda contiene factores intracelulares con funciones clave en el sistema nervioso.
Según la hipótesis de “menos es más”, estas dos subfamilias podrían representar el conjunto mínimo de FGFs necesario en un cordado, mientras que la pérdida de las otras seis habría contribuido a la evolución de rasgos únicos en las apendicularias. Por ejemplo, en otros tunicados, las subfamilias Fgf7/10/22 y Fgf8/17/18 regulan la metamorfosis, un proceso en el que las larvas, inicialmente nadadoras gracias a su cola, la pierden y se transforman en adultos sésiles. La pérdida de estas subfamilias en las apendicularias podría explicar por qué conservan la cola y mantienen un estilo de vida libre durante toda su existencia.
Nueva complejidad
La mayor sorpresa fue descubrir que, además de ser las únicas supervivientes, las subfamilias Fgf9/16/20 y Fgf11/12/13/14 se han expandido mediante duplicaciones génicas alcanzando un total de 10 genes. Al analizar su estructura, las proteínas que codifican y sus patrones de expresión, observamos que cada gen ha adquirido funciones únicas. En algunos casos, estas funciones se relacionan con innovaciones evolutivas propias de las apendicularias; en otros, los genes duplicados habrían asumido el papel de las subfamilias perdidas. En cualquier caso, nuestros resultados indican que los genes resultantes de esta expansión se han diversificado funcionalmente.
Así, la pérdida de la mayoría de subfamilias de FGF, combinada con la expansión de las dos supervivientes, parece explicar algunas de las adaptaciones evolutivas características de O. dioica. Este patrón coincide con otro hallazgo llamativo: a pesar de la drástica reducción de su genoma, O. dioica posee más de 20 000 genes, una cifra comparable a la del ser humano y superior a la de otros tunicados con genomas más grandes.
Menos, pero más
stro
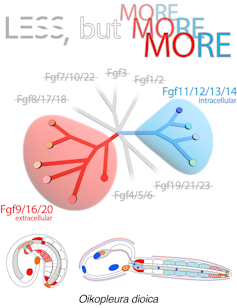
La evolución de la vía de FGF en las apendicularias respalda la hipótesis de “menos es más”: la pérdida de genes ha facilitado su adaptación evolutiva. Sin embargo, lejos de tratarse de una simple reducción, la eliminación de genes ha ido acompañada de la expansión y diversificación de los que se conservaron, generando una “nueva complejidad”.
Este fenómeno es similar a lo observado en la evolución de los receptores olfativos en animales adaptados a distintos entornos o en la diversificación de las proteínas globinas en mamíferos, lo que sugiere un patrón recurrente en la naturaleza.
A partir de estos hallazgos, proponemos un modelo que amplía la hipótesis de “menos es más” a contextos en los que la pérdida génica ocurre junto con la duplicación y diversificación de los genes supervivientes. Lo llamamos “menos, pero más”, para destacar cómo la pérdida de genes puede abrir nuevas oportunidades evolutivas a través de la expansión funcional de los restantes.
Este modelo identifica cuatro consecuencias principales: la conservación de funciones ancestrales, la pérdida de funciones asociadas a los genes eliminados, el intercambio de funciones entre genes duplicados y genes perdidos y, por último, la aparición de nuevas funciones tras las duplicaciones.
Lejos de constituir un proceso meramente simplificador, la pérdida génica puede actuar como un motor evolutivo al influir en la reestructuración del genoma y promoviendo la innovación y la adaptación.
Cristian Cañestro recibe fondos de PID2019-110562GB-I00 y PID2022-141627NB-I00 del Ministerio Español de Ciencia, Innovación y Universidades/Agencia Estatal de Investigación/10.13039/501100011033/ y FEDER/UE; ICREA Acadèmia Ac2215698 y 2021-SGR00372 AGAUR, Generalitat de Catalunya.
Gaspar Sánchez Serna recibió fondos del Ministerio de Ciencia, Innovación y Universidades del Gobierno de España (2019-2023).
